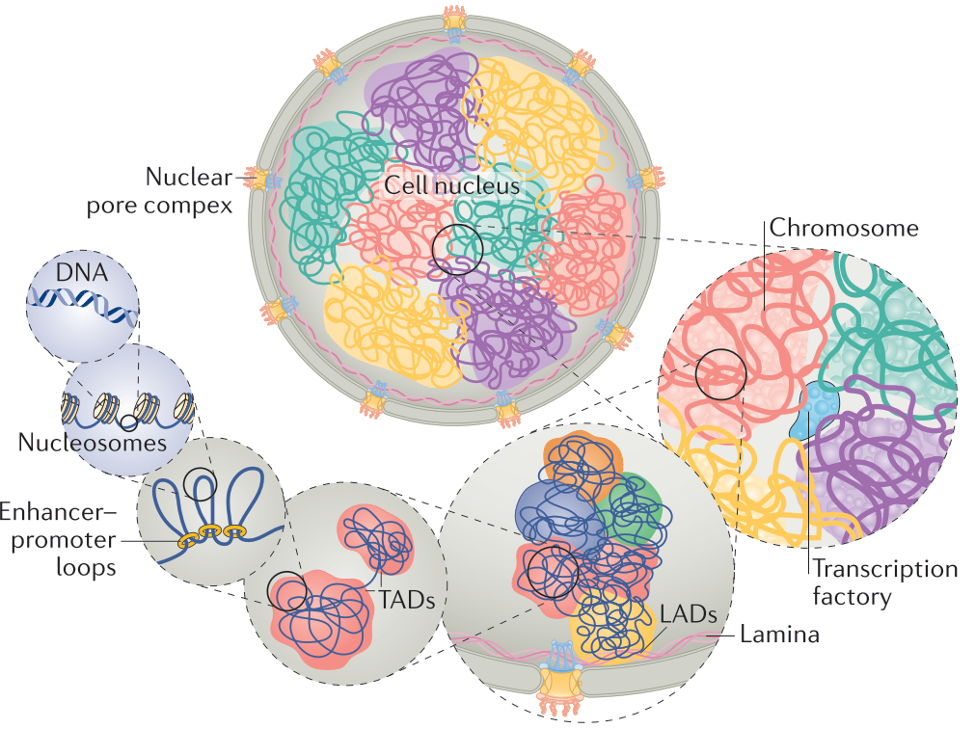
L’invenzione propone un metodo per rilevare i cambiamenti della cromatina, in particolare dell’eterocromatina il cui stato e accessibilità è indicatore dello stato di salute della cellule o della presenza di specifiche malattie e tumori. Pertanto, metodi affidabili per caratterizzare l’eterocromatina e le sue alterazioni sono essenziali per la comunità scientifica biomedica.